Identifizierung und Kartierung des Genoms
Genotypisierung:
Unter dem Begriff Genotypisierung versteht man die Anfertigung eines
genetischen Profiles, eines genetischen Fingerabdruckes, der eine eindeutige
Zuordnung zulässt, wie etwa: Aus welcher Zucht stammt dieses Tier,
von welchem Saatgut stammt diese Pflanze oder von welchem Menschen stammt
ein Bluttropfen.
Diese molekulargenetische Identifikation kann nicht nur für phylogenetische
Fragestellungen verwendet werden, diese Stammbaumanalysen sind beispielsweise
für Zoos von Interesse. Es können Inzuchteffekte vermieden werden,
und für die Wissenschaft wichtige Fragestellungen erfahren dadurch
eine Klärung. Der Genetische Fingerabdruck erlaubt auch den Herkunftsnachweis.
Eine gerade durch mehrere Skandale sensibilisierte Bevölkerung wird
verstärkt wissen wollen, woher die Nahrungsmittel kommen und DNA-Analysen
sind das Mittel der Wahl, um rasch und effizient diese dringenden Fragen
zu beantwor-ten.
Minisatelliten-Analyse (Southern Blot):
Das wichtigste Werkzeug bei der Southern Blot-Analyse von RFLP-Systemen
sind bestimmte Eiweißstoffe, Restriktionsenzyme genannt, die aus
Bakterien isoliert werden, und die wie chemische Scheren die DNA an ganz
bestimmten Stellen schneiden können.
"RFLP" bedeutet "restriction fragment length polymorphism".
Gemeint ist, dass jeder Mensch auf der Erde ein ganz spezielles Muster
an DNA-Fragmentlängen aufweist.
Als Erkennungsmuster für diese Enzyme dienen bestimmte Basensequenzen,
die mehr oder weniger zufällig verstreut auf dem DNA-Molekül
vorkommen. Wenn nun die DNA in den flankierenden Bereichen von Minisatellitensequenzen
geschnitten wird, so entstehen DNA-Fragmente, deren Länge abhängig
ist von der Anzahl der Minisatelliten-Wiederholungseinheiten. Bei der
Southern-Blot-Analyse werden DNA-Fragmente, die durch Behandlung mit den
schneidenden Enzymen entstanden sind, in einer Elektrophorese nach ihrer
Länge getrennt, auf eine Membran übertragen und mittels radioaktiv
oder Enzym-markierter "DNA-Sonden" die nur die gesuchten Fragmente
erkennen können, sichtbar gemacht. So entstehen RFLP-Muster, deren
Zusammensetzung typisch für ein einzelnes Individuum ist. Die beiden
Merkmale eines jeden Genortes lassen sich dabei jeweils von der Mutter
bzw. vom Vater ableiten.
PCR
Der Nachteil der DNA-Analysetechnik ist die Menge benötigter DNA
(5-10 µg). Das ist bei Vaterschaftsfällen kein Problem. Bei
winzigen Blut-, Sperma oder Speichelspuren liegt jedoch eine nicht überwindbare
Mengenbeschränkung vor.
Daher nutzen die Labors die Methode der DNA-Vervielfältigung durch
die Po-lymerasekettenreaktion (polymerase chain reaction, PCR), für
deren Entwicklung KARY MULLIS 1993 den Nobelpreis erhielt. Durch die Wahl
geeigne-ter DNA-Startermoleküle wird bei der PCR-Typisierung nicht
das gesamte Genom zerschnitten und aufgetrennt, sondern es werden ausgewählte
DNA-Strecken vervielfältigt.
Analyse mittels PCR
PCR ist ein in vitro (Reagenzglas-) Verfahren zur gezielten Vermehrung
von DNA mit Hilfe von synthetischen Oligonukleotiden und dem Enzym DNA-Polymerase.
Durch wiederholte Zyklen wird aus einer DNA-Lösung ein spezifi-scher
DNA-Abschnitt vermehrt.
Synthetische Oligonukleotide (eine nur kurze Reihe von Basen auf der
DNA) definieren den DNA-Abschnitt, der vervielfältigt werden soll.
Sie werden PCR-Primer genannt, da nach ihrer Anlagerung an einen DNA-Einzelstrang
von ihrem Ende ausgehend das Enzym DNA-Polymerase den komplementären
Strang neu synthetisieren kann.
Die Primer für ein bestimmtes PCR-System werden so gewählt,
dass sie den zu untersuchenden Abschnitt der DNA einschließen. Damit
die DNA-Neusynthese stattfinden kann, müssen neben den bereits erwähnten
Primern und der Polymerase auch die vier verschiedenen reaktiven DNA-Bausteine,
die so genannten Nukleotide, im Reaktionsgemisch vorhanden sein.
Die eigentliche PCR-Reaktion findet in kleinen Reaktionsgefäßen
in einem so genannten Thermocycler statt.
Am Beginn jedes PCR-Zyklus steht eine Denaturierung der Doppelstrang-DNA
durch Erwärmung auf eine Temperatur über 90°C, wodurch die
beiden komplementären DNA-Stränge voneinander getrennt werden.
Als nächster Schritt wird das Reaktionsgemisch auf eine für
die beiden Primer spezifische Temperatur zwischen 50°C und 65°C
abgekühlt. Bei dieser Temperatur könnten sich die PCR-Primer
an die DNA-Einzelstränge anlagern. Die Temperatur muss so gewählt
werden, dass sich die Primer nur dort anla-gern, wo ihre Sequenz vollständig
mit der des DNA-Einzelstrangs übereinstimmt.
Im dritten Schritt des PCR-Zyklus, bei ca. 72°C, findet schließlich
ausgehend von den Enden der Primer eine komplementäre Ergänzung
der DNA-Einzelstränge statt, wodurch in diesem Bereich ein DNA-Doppelstrang
entsteht.
In jedem Folgezyklus findet nunmehr erneut eine Verdopplung dieses DNA-Abschnitts
statt, wodurch es zu einer exponentiellen Vermehrung der Zielsequenz kommt.
Typischerweise werden bei PCR-Vermehrung 28 - 32 Zyklen durchgeführt.
Daraus errechnet sich theoretisch ausgehend von einer einzel-nen Kopie
zu Beginn der PCR, dass am Ende nach 32 Zyklen 2 Milliarden Kopien des
zu untersuchenden DNA-Abschnittes vorliegen. Tatsächlich kann man
jedoch nicht mit einer 100%igen Verdopplung pro Zyklus rechnen. Realistische
Werte liegen zwischen 70 und 90 %. Bei einer durchschnittlichen Effizienz
von 80% errechnen sich nach 32 Zyklen 5 Mio. Kopien der Ausgangskopie
der DNA. Dies reicht für eine Analyse mittels Elektrophorese.
Kapillar-Elektrophorese:
In der Gel - Elektrophorese wandern geladene, biologische Moleküle
im elektrischen Feld. Die in Protonen und konjugierte Basen zerfallene
DNA wandert vom negativen zum positiven Pol. Je nach Form und Größe
wandern die die kleineren Moleküle, die die Gelporen schneller passieren,
schneller als Größere.
Die Analyse der PCR-Produkte, welche sich durch die Anwesenheit von Mikrosatelliten-Polymorphismen
in ihrer Länge unterscheiden, erfolgt durch eine automatische Fragmentanalyse
mittels Kapillar-Elektrophorese.
Die DNA-Fragmente mit den Repeat-Merkmalen werden an der Kathode (-)
auf-getragen und wandern bei Anlegen einer Hochspannung durch ihre eigene
negative Ladung in die Kapillare in Richtung Anode (+) ein. In der Kapillare
befindet sich ein flüssiges Polymer, dass wie ein Sieb die Fragmente
sortiert und bei fortschreitender Wanderung durch die Kapillare auftrennt.
Die PCR-Produkte (DNA-Teile) werden durch die Verwendung jeweils eines
PCR-Primers für jedes System sichtbar gemacht, der mit einem Fluoreszenzfarbstoff
(blau, grün oder gelb) markiert worden ist. Weiterhin wird ein mit
rotem Farbstoff markierter interner DNA-Längenstandard zugefügt,
der zur Messung der PCR-Produkte unbekannter Fragmentlänge benötigt
wird. Wandern nun die markierten DNA-Fragmente der zu untersuchenden Probe
aufgrund der Polarität des elektrischen Feldes der Elektrophorese
durch das in der Kapillare befindliche gelartige Polymer von der Kathode
zur Anode, werden sie der Länge nach aufgetrennt, d.h. die kleinen
Fragmente können schneller durch das Gel dringen als die großen
Fragmente. Sobald diese Fragmente am Fenster des Detektors vorbeiwandern,
werden die Farbstoffmoleküle durch einen Laser-strahl zur Fluoreszenz
angeregt. Diese Fluoreszenz wird nun durch ein optisches System auf einen
Detektor mit CCD-Kamera gebracht und dort nach Wellenlänge und Signalstärke
in digitale Messwerte umgewandelt.
Diese Werte werden schließlich mittels EDV-Analyse mit den bekannten
Fragmentlängen des internen Standards verglichen und dann den Allelen
der einzelnen DNA-Systeme gestaffelt nach Farbmarkierung und Fragmentlänge
zugeordnet.
Abb.: Beispiel für DNA-Fragmente, die durch Elektrophorese getrennt
wurden.
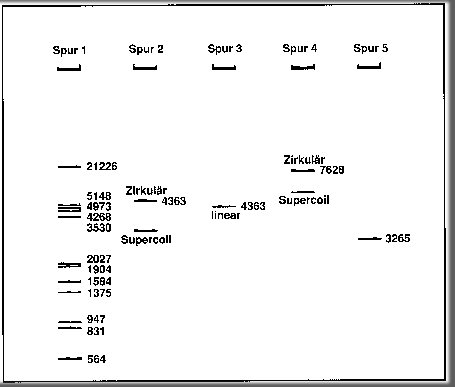 Spur
1 zeigt die Banden der DNA-Fragmente des DNA-Markers III, der durch
Schneiden des DNA-Fadens des Bakteriophagen Lambda (Lambda-DNA) mit dem
Restriktionsen-zymen Eco RI und Hind III entstanden ist. Den Banden sind
jeweils die Basenpaare (bp) der Fragmente zugeordnet. Spur
1 zeigt die Banden der DNA-Fragmente des DNA-Markers III, der durch
Schneiden des DNA-Fadens des Bakteriophagen Lambda (Lambda-DNA) mit dem
Restriktionsen-zymen Eco RI und Hind III entstanden ist. Den Banden sind
jeweils die Basenpaare (bp) der Fragmente zugeordnet.
Spur 2 zeigt die Bande der ungeschnittenen DNA des Plasmids pBR322
(p=Plasmid, BR = Bolivar und Rodrigues, die 1977 das Plasmid entwickelten,
3 = Plasmid besteht aus drei Teilen, 20 = in einem Bakterium können
bis zu 20 Plasmide kopiert werden, 2 = 2 Gene für Antibiotikaresistenzen)
Spur 3 zeigt die Bande des Restriktionsansatzes: den mit Nhe I
einmal aufgeschnitte-nen Plasmidring von pBR322, die jetzt linear vorliegende
DNA. Das Restriktionsenzym NheI wurde aus Neisseria mucosa subspecies
heidelbergensis isoliert.
Spur 4 die Bande des Plasmids pBR322 lacZ+, Plasmidring pBR322
mit eingebautem lacZ+-Gen. Die DNA kommt dabei in zwei Zuständen
vor: als Supercoil-Plasmid und als zirkulärer Plasmid. Die verdrillte
Supercoilform durchdringt das Gel schneller als die lineare und die zirkuläre
DNA.
(Aus: http://www.uni-koblenz.de/~odsgroe/dnaanaly.php)
|


